
【环球网科技归纳报导】3月12日音讯,中国科学院主动化研讨所多模态AI体系实验室杨戈团队与中国科学院生物物理研讨所蛋白质科学研讨渠道生物成像中心孙飞团队协作,以AI技能赋能原位结构生物学,提出了一种依据弱监督深度学习的快速精确颗粒选择办法DeepETPicker。
生物大分子(如蛋白质)的结构与功用会跟着细胞生理状况的改变不断进行动态调整。原位结构生物学是在挨近天然生理状况下研讨生物大分子结构和功用的科学,而原位冷冻电镜技能(cryo-electron tomography, Cryo-ET)以其高分辨率和在挨近生理条件下调查样品的特色,成为原位结构生物学研讨中的要害手法。原位冷冻电镜的技能流程触及样品制备、数据收集、电子断层重建、颗粒选择、粒子平平等多个进程。生物大分子的颗粒选择,即定位辨认,是其间一个要害环节。受限于Cryo-ET图画的极低信噪比和重建伪影等要素,不计其数个方针颗粒的手动选择极为耗时吃力,现有主动选择办法的运用遭到人工标示量高、计算成本高和颗粒质量不抱负等多方面约束。
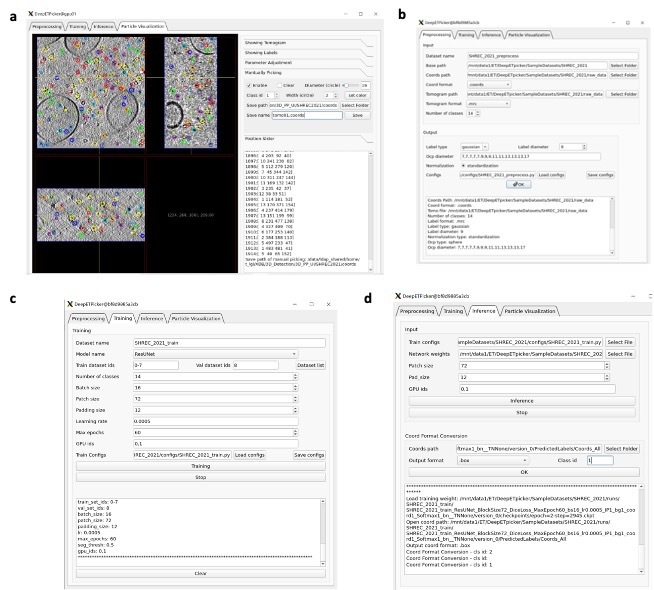
DeepETPicker仅需求少数人工标示颗粒进行练习就可以完结快速精确三维颗粒主动选择。为下降对人工标示量的需求,DeepETPicker优选简化标签来替代实在标签,并选用了更高效的模型架构、更丰厚的数据增强技能和堆叠分区战略来进步小练习集时模型的功能;为进步颗粒定位的速度,DeepETPicker选用图形处理器(GPU)加快的均匀池化-非极大值按捺(MP-NMS, mean pooling and non-maximum suppression)后处理操作,与现有的聚类后处理办法比较进步选择速度数十倍。此外,为便利用户运用,项目团队推出了操作简练、界面友爱的开源软件(图1)以辅佐用户完结图画预处理、颗粒标示、模型练习与推理等操作。
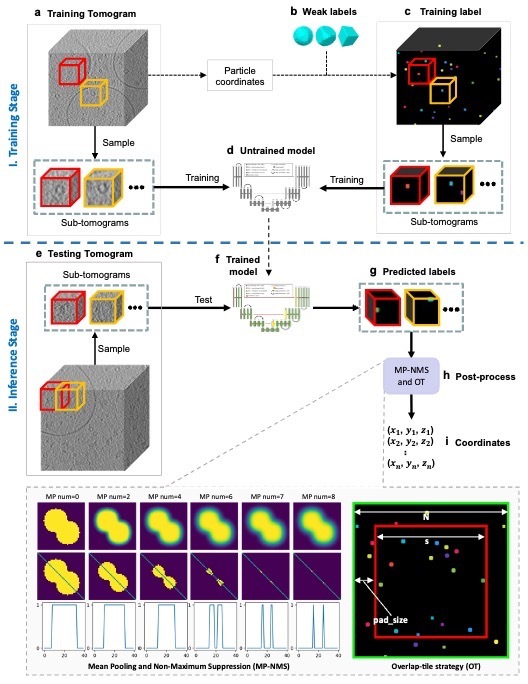
运用DeepETPicker从冷冻电子断层扫描图画中选择颗粒的全体作业流程如图2所示,包含练习阶段(图2a-c)和推理阶段(图2e-g)。在练习数据的预备阶段,研讨人员优选了弱标签TBall-M来替代实在掩模以减轻人工标示担负,并在模型架构规划方面,引进坐标卷积(coordinated convolution)和图画金字塔(image pyramid inputs)到3D-ResUNet的切割架构中以进步定位的精确性。在模型推理阶段,DeepETPicker选用堆叠断层图分区战略(OT, overlap-tile),避免了因为边际体素切割精度欠安而发生的负面影响,从而结合MP-NMS操作加快了颗粒中心定位进程。
研讨团队将DeepETPicker与现在功能最优的颗粒选择办法在多种冷冻电子断层扫描数据集进步行了功能点评比照,选用六个定量目标全面点评颗粒选择的质量(图3):精确率-召回率(Precision-Recall)、F1-分数(F1-score)、对数似然概率贡献度(Log-likelihood Contribution)、最大值概率(maximum value probability)、RH分辨率(Rosenthal-Henderson resolution)、大局分辨率。依据成果得出,DeepETPicker在仿真与实在数据集上均可完结快速精确的颗粒选择,其归纳功能显着优于现有的其他办法,生物大分子结构重建到达的分辨率也到达选用专家人工选择颗粒进行结构重建相同的水平,这进一步表现了DeepETPicker在原位高分辨率结构解析中的实用价值。DeepETPicker将为选用原位冷冻电镜技能的原位结构生物学研讨供给有力的支撑。